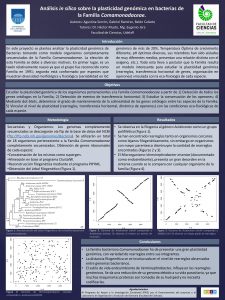
 Integrantes: Belén Calvete, Agustina Serrón, Gabriel Ramírez Docentes orientadores: Dr. Héctor Musto, Mg. Eugenio Jara Servicio: Facultad de Ciencias, UdelaR. En este proyecto se analizó la plasticidad genómica de Bacterias, tomando como modelo organismos completamente secuenciados en la Familia Comamonadaceae. Para esto se realizó la detección de todos los genes ortólogos en la Familia. Mediante dot-blots, se determinó el grado de mantenimiento de la colinearidad de los genes ortólogos entre las especies de la Familia. Por último, a partir de estos datos vinculamos el nivel de rearreglos con las condiciones eco-fisiológicas de cada especie. Esta Familia se eligió por presentar una gran diversidad morfológica y fisiológica (variabilidad en GC genómico de más de 20%, Temperatura Optima de crecimiento diferente, pH óptimos diversos, sus miembros han sido aislados de diferentes medios, presentan distintos tipos de respiración, etc.). Esto lleva a postular que la Familia Comamonadaceae resulta un grupo interesante para estudiar la plasticidad genómica vinculada con la eco-fisiología de cada especie. Se han encontrado rearreglos, tanto en organismos cercanos como lejanos. Pero se ha demostrado que los rearreglos presenten una disminución en organismos cercanos, aumentando cuando se compara con dos organismos lejanos filogenéticamente. Es de destacar que el organismo Verminefphrobacter presenta un gran desorden en la sintenia cuando se le compara con cualquier organismo de toda la familia. Asumimos que estos rearreglos se han debido a su estilo de vida parasitaria. La dinámica genómica es un patrón importante para poder entender procesos evolutivos desde un punto de vista genómico, y aún falta para poder entender los factores que determinan los cambios en los genomas procariotas.
Integrantes: Belén Calvete, Agustina Serrón, Gabriel Ramírez Docentes orientadores: Dr. Héctor Musto, Mg. Eugenio Jara Servicio: Facultad de Ciencias, UdelaR. En este proyecto se analizó la plasticidad genómica de Bacterias, tomando como modelo organismos completamente secuenciados en la Familia Comamonadaceae. Para esto se realizó la detección de todos los genes ortólogos en la Familia. Mediante dot-blots, se determinó el grado de mantenimiento de la colinearidad de los genes ortólogos entre las especies de la Familia. Por último, a partir de estos datos vinculamos el nivel de rearreglos con las condiciones eco-fisiológicas de cada especie. Esta Familia se eligió por presentar una gran diversidad morfológica y fisiológica (variabilidad en GC genómico de más de 20%, Temperatura Optima de crecimiento diferente, pH óptimos diversos, sus miembros han sido aislados de diferentes medios, presentan distintos tipos de respiración, etc.). Esto lleva a postular que la Familia Comamonadaceae resulta un grupo interesante para estudiar la plasticidad genómica vinculada con la eco-fisiología de cada especie. Se han encontrado rearreglos, tanto en organismos cercanos como lejanos. Pero se ha demostrado que los rearreglos presenten una disminución en organismos cercanos, aumentando cuando se compara con dos organismos lejanos filogenéticamente. Es de destacar que el organismo Verminefphrobacter presenta un gran desorden en la sintenia cuando se le compara con cualquier organismo de toda la familia. Asumimos que estos rearreglos se han debido a su estilo de vida parasitaria. La dinámica genómica es un patrón importante para poder entender procesos evolutivos desde un punto de vista genómico, y aún falta para poder entender los factores que determinan los cambios en los genomas procariotas.

![[CURLN] MORFOGÉNESIS DE ESPECIES NATIVAS DE CAMPO NATURAL](https://www.estudiantes.csic.edu.uy/wp-content/uploads/2017/05/232_archivoarchivo-500x383.jpg)
![[IENBA] Estudio de los aportes de la lectura y escritura en la formación universitaria a través del análisis del curso “Estéticas Contemporáneas” del Instituto ‘Escuela Nacional de Bellas Artes’ de la Universidad de la República.](https://www.estudiantes.csic.edu.uy/wp-content/uploads/2017/05/170_archivoarchivo-500x383.jpg)
![[CURLN] Evaluación de antixenosis y tolerancia de de Schizaphis graminum (Hemiptera: Aphididae) en cebada.](https://www.estudiantes.csic.edu.uy/wp-content/uploads/2017/05/PAIE-Tagliani-500x383.jpg)
![[IENBA] Cuerpo epistemológico. Estudios sobre el cuerpo en las prácticas universitarias](https://www.estudiantes.csic.edu.uy/wp-content/uploads/2017/05/139_archivoarchivo-500x383.jpg)
![[CURLN] EFECTOS DE LA CF EN EL NIVEL SILÁBICO DE LA ESCRITURA Y EL ACCESO A REDES SEMÁNTICAS](https://www.estudiantes.csic.edu.uy/wp-content/uploads/2017/05/100_archivoarchivo-500x383.jpg)